ポアソン分布のパラメーター平均 λ だけからなる。Stan では poisson 関数を使ってモデルを構築することができる。ここで、λ = 20 であるポアソン分布から乱数を 100 個生成して、次に Stan で乱数が生成されたポアソン分布のパラメーターを推定する例を示す。
x <- rpois(n = 100, lambda = 20)Stan を利用して x を生成したポアソン分布のパラメーター λ を求める。Stan コードの data ブロックには、これから入力する値である x を定義する。Stan 自身は、x に何個のデータが入っているのかを計算できないので、外部から与える必要がある。このため、x の他に、x の個数も data ブロックで定義する。次に、parameters ブロックには、これから推定したいポアソン分布のパラメーターである λ を定義する。最後に、model ブロックで、入力値変数とパラメーター変数を使ってポアソン分布モデルを構築する。
data {
int n;
int x[n];
}
parameters {
real lambda;
}
model {
x ~ poisson(lambda);
}R では、次のようにして Stan コードを呼び出して実行する。推定結果を見ると、λ は 19.93 に推定され、実際に利用した 20 に近いことがわかる。
d <- list(x = x, n = length(x))
fit <- stan(file = 'poissondisp.stan', data = d)
fit
## Inference for Stan model: tmp.
## 4 chains, each with iter=2000; warmup=1000; thin=1;
## post-warmup draws per chain=1000, total post-warmup draws=4000.
##
## mean se_mean sd 2.5% 25% 50% 75% 97.5% n_eff Rhat
## lambda 19.93 0.01 0.45 19.07 19.62 19.94 20.23 20.81 1544 1
## lp__ 3973.00 0.01 0.67 3971.09 3972.81 3973.27 3973.45 3973.50 2115 1各回 MCMC サンプリングの結果をプロットする場合は、stan_trace 関数を利用し、パラメーターの事後確率のヒストグラムをプロットする場合は stan_hist 関数を利用する。
stan_trace(fit)
stan_hist(fit)
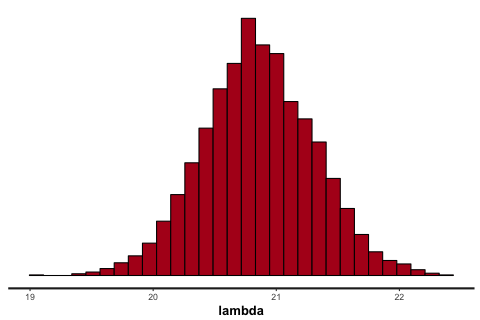
MCMC サンプリング結果は、そのままパラメーターが取りうる値とみなすことができる。そのため、α/2 分位点から 1 - α/2 分位点までの区間は、そのパラメーターにおける (1 - α)% のベイズ信用区間とみなすことができる。このことを利用して、平均と標準偏差の 95% 信用区間を求めるには、次のように計算すればいい。
ms <- rstan::extract(fit)
quantile(ms$lambda, probs = c(0.025, 0.975))
## 2.5% 97.5%
## 19.06668 20.80618