このページでは、Stan を使って個体差を考慮した単回帰モデルを構築する方法を示す。サンプルデータとして R に標準実装されている Orange とよばれるデータセットを使用する。このデータセットは、個体番号(Tree)、樹齢(age)、周長(circumference)の 3 列からなる。ここで、樹齢を使って周長を説明するモデルを構築することを目的とする。
data(Orange)
head(Orange)
## Tree age circumference
## 1 1 118 30
## 2 1 484 58
## 3 1 664 87
## 4 1 1004 115
## 5 1 1231 120
## 6 1 1372 142
plot(Orange$age, Orange$circumference, col = Orange$Tree)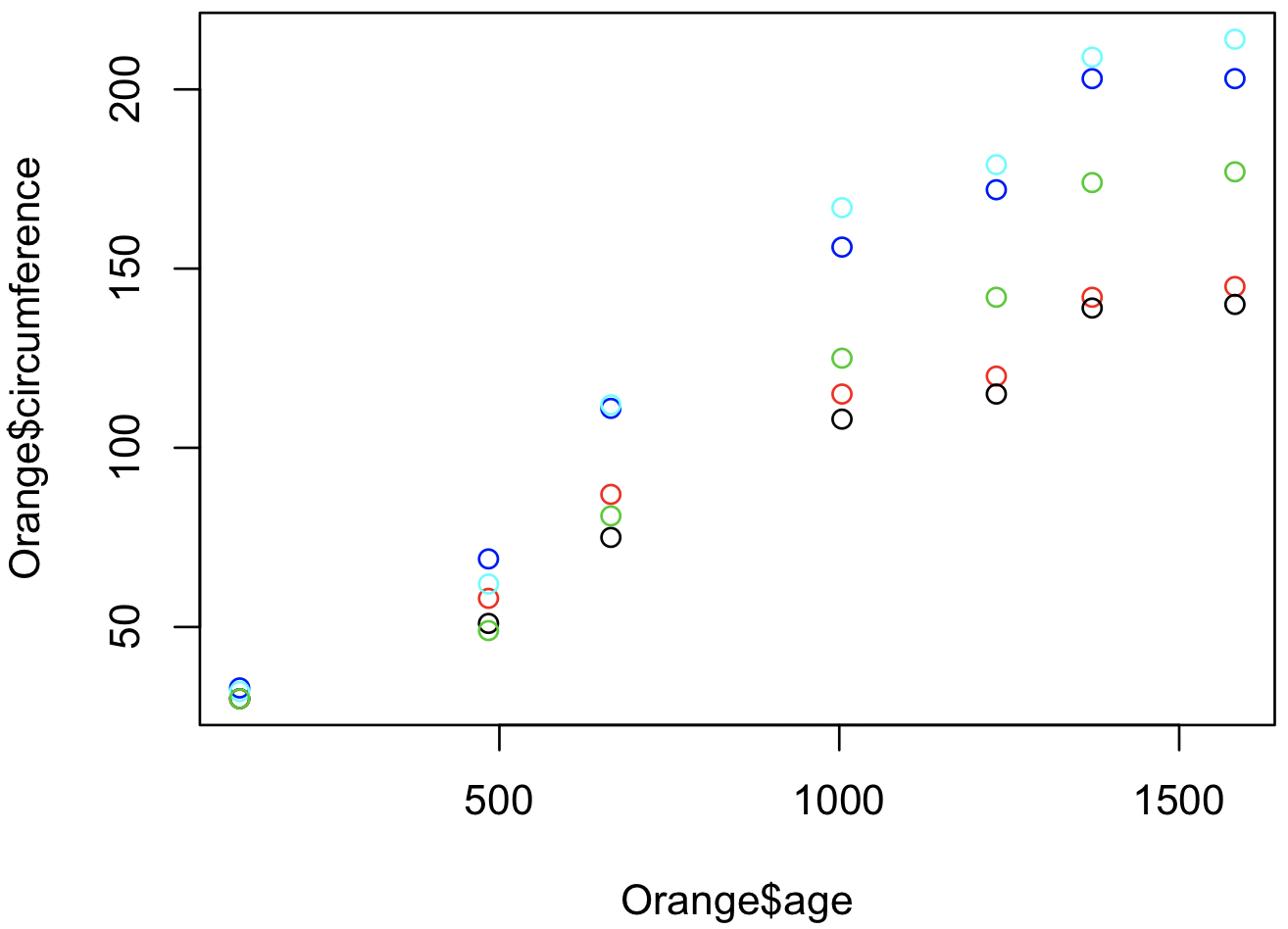
Stan モデル
Stan でモデルを記述していく。Orange データセットには 5 つの個体のデータが入っている。個体差を考慮に入れたモデルを構築したいので、まず個体差を表すパラメーターを導入する。全個体の(線型予測子の)平均を beta_ave とおいたとき、個体 1〜5 の個体差は平均からどれぐらい離れているかで表すことができる。その差をパラメーターとして定義する。parameters ブロックでこれらのパラメーターを定義する。また、周長 y をサンプリングするために、平均だけでは不十分であるので、分散も必要である。そのため、パラメーターとして分散も parameters ブロックに加える。
平均と分散が用意できので、個体 i の周長 y は次にようにサンプリングすることができる。
\[ y \sim Normal\left( \mu_{i}, \sigma \right) \]個体 i の平均は、個体差を考慮するので、次のように計算される。
\[ \beta_{0}^{(i)} = \beta_{0}^{overall} + \beta_{0}^{i\_diff\_from\_all} \] \[ \beta_{1}^{(i)} = \beta_{1}^{overall} + \beta_{1}^{i\_diff\_from\_all} \] \[ \mu_{i} = \beta_{0}^{(i)} + \beta_{1}^{(i)}x \]このように、正規分布で使用する μi は、parameters ブロックで定義した β などではない。そのため、transformed parameters ブロックで、β などのパラメーターを平均 μ に変換する命令を記述する。
また、パラメーターを推定した後に、各個体について樹齢が 0〜1600 までの予測区間を計算したいので、generated quantities で推定されたパラメーターを使って再サンプリングを行なうように記述する。
data {
// data points
int N;
real x[N];
real y[N];
// number of trees and tree ID information
int T;
int tree[N];
// tree ages for prediction
int new_N;
real new_x[new_N];
}
parameters {
// parameters for calculating the overall average
real beta0_ave;
real beta1_ave;
// differences from the overall averages for each tree
real beta0_diff[T];
real beta1_diff[T];
// standard deviation for sampling `y`
real<lower=0> sigma;
}
transformed parameters {
real beta0[T];
real beta1[T];
real mu[N];
for (t in 1:T) {
beta0[t] = beta0_ave + beta0_diff[t];
beta1[t] = beta1_ave + beta1_diff[t];
}
for (n in 1:N) {
mu[n] = beta0[tree[n]] + beta1[tree[n]] * x[n];
}
}
model {
for (n in 1:N) {
y[n] ~ normal(mu[n], sigma);
}
}
generated quantities {
real muhat[new_N, T];
real yhat[new_N, T];
for (t in 1:T) {
for (n in 1:new_N) {
muhat[n, t] = beta0[t] + beta1[t] * new_x[n];
yhat[n, t] = normal_rng(muhat[n, t], sigma);
}
}
} パラメーター推定
次に R の rstan パッケージを利用して、パラメーター推定を行う。
library(rstan)
new.x <- seq(0, 1600, 10)
x <- Orange$age
y <- Orange$circumference
treeid <- as.integer(Orange$Tree)
d <- list(x = x, y = y, N = length(x), tree = treeid, T = length(unique(treeid)),
new_x = new.x, new_N = length(new.x))
fit <- stan(file = 'lm.stan', data = d)上のコードを実行すると、実行結果が fit オブジェクトに保存される。このオブジェクトには様々な情報が含まれ、そのまま表示すると非常に見づらい。ここでは、5 つの木それぞれのパラメーター β0 および β1 の 95% 信頼区間を見てみる。
ms <- rstan::extract(fit, pars = c('beta0', 'beta1'))
apply(ms$beta0, 2, quantile, probs = c(0.025, 0.500, 0.975))
## [,1] [,2] [,3] [,4] [,5]
## 2.5% 2.316493 6.720595 -9.149217 2.716527 -4.20210
## 50% 19.610563 24.550709 8.398044 19.193066 15.75030
## 97.5% 36.404058 40.306671 24.462012 34.905749 32.02995
apply(ms$beta1, 2, quantile, probs = c(0.025, 0.500, 0.975))
## [,1] [,2] [,3] [,4] [,5]
## 2.5% 0.06455640 0.06534071 0.09601257 0.1099995 0.1183769
## 50% 0.08088153 0.08150999 0.11128441 0.1256886 0.1343835
## 97.5% 0.09763204 0.09872538 0.12810771 0.1421530 0.1525764
stan_hist(fit, pars = c('beta0', 'beta1'))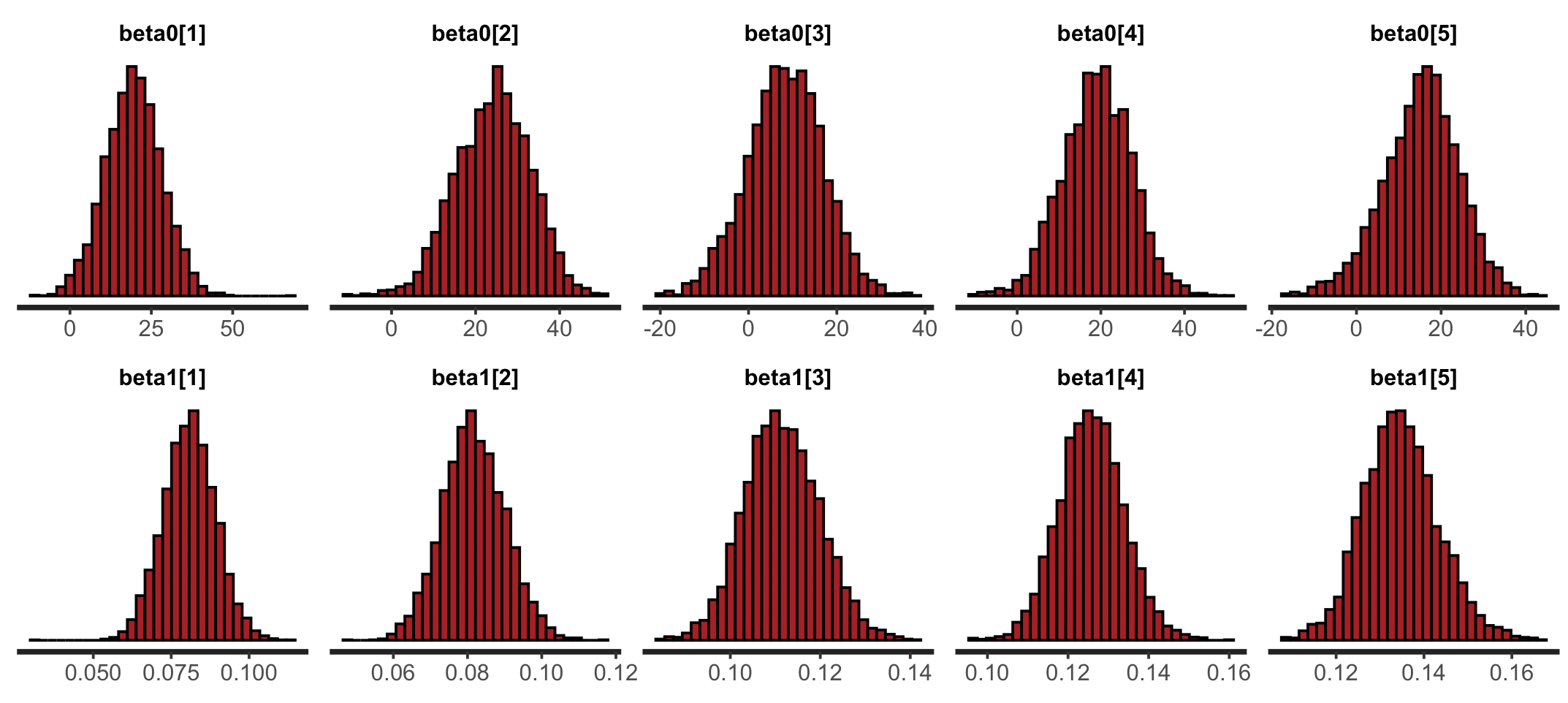
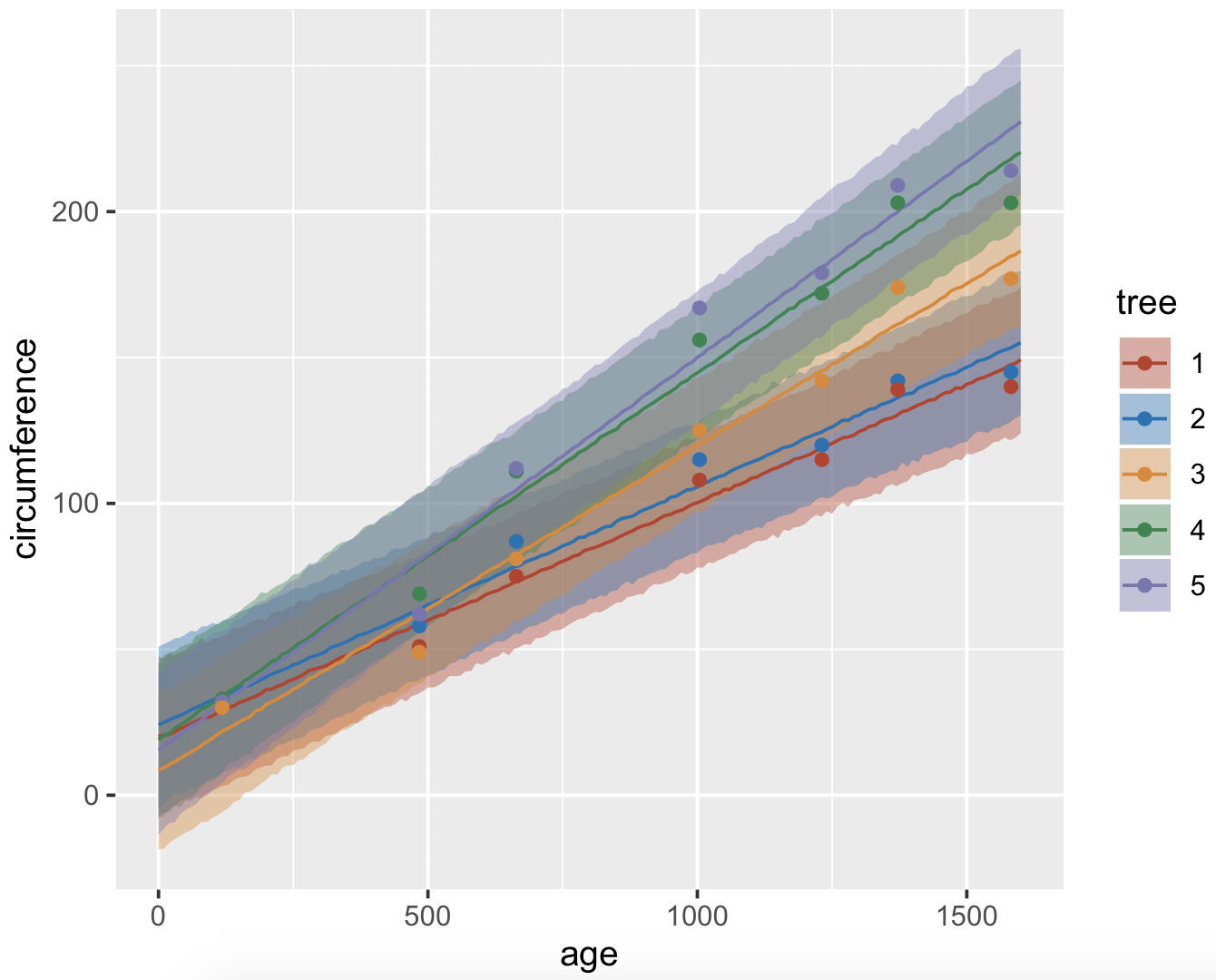
次に Stan コードの generated quantities のブロックで再サンプリングしたデータを用いて、個体ごとの予測区間を描く。
ms <- rstan::extract(fit, pars = c('yhat'))
dim(ms$yhat)
df <- NULL
for (i in 1:5) {
df <- rbind(df,
data.frame(x = new.x, tree = i,
lower = apply(ms$yhat[, , i], 2, quantile, prob = 0.025),
median = apply(ms$yhat[, , i], 2, quantile, prob = 0.500),
upper = apply(ms$yhat[, , i], 2, quantile, prob = 0.975)))
}
df$tree <- as.factor(df$tree)
library(ggplot2)
library(ggsci)
g <- ggplot(df, aes(x = x))
g <- g + geom_ribbon(aes(ymin = lower, ymax = upper, fill = tree), alpha = 0.4)
g <- g + geom_line(aes(y = median, color = tree))
g <- g + geom_point(data = data.frame(x = d$x, y = d$y, tree = as.factor(treeid)),
aes(x = x, y = y, colour = tree))
g <- g + scale_color_nejm() + scale_fill_nejm()
g <- g + xlab('age') + ylab('circumference')
print(g)