このページでは、Stan を使用して簡単な回帰モデルのパラメーターを推定する例を示す。ここで、サンプルデータとして、trees と呼ばれているデータセットを使用する。このデータセットには 31 本の桜の木の周長(Grith)、高さ(Height)、および容積(Volume)のデータが記録されている。樹木が高くなるには、それを支えるために幹も太くなる必要がある。そのため、幹が太さを用いて、木の高さを推定できるかもしれない。そこで、この仮定をもとに、Grith を説明変数とし、Height を応答変数として、回帰モデルを構築する。
data(trees)
head(trees)
## Girth Height Volume
## 1 8.3 70 10.3
## 2 8.6 65 10.3
## 3 8.8 63 10.2
## 4 10.5 72 16.4
## 5 10.7 81 18.8
## 6 10.8 83 19.7
library(ggplot2)
g <- ggplot(trees, aes(x = Girth, y = Height)) + geom_point()
print(g)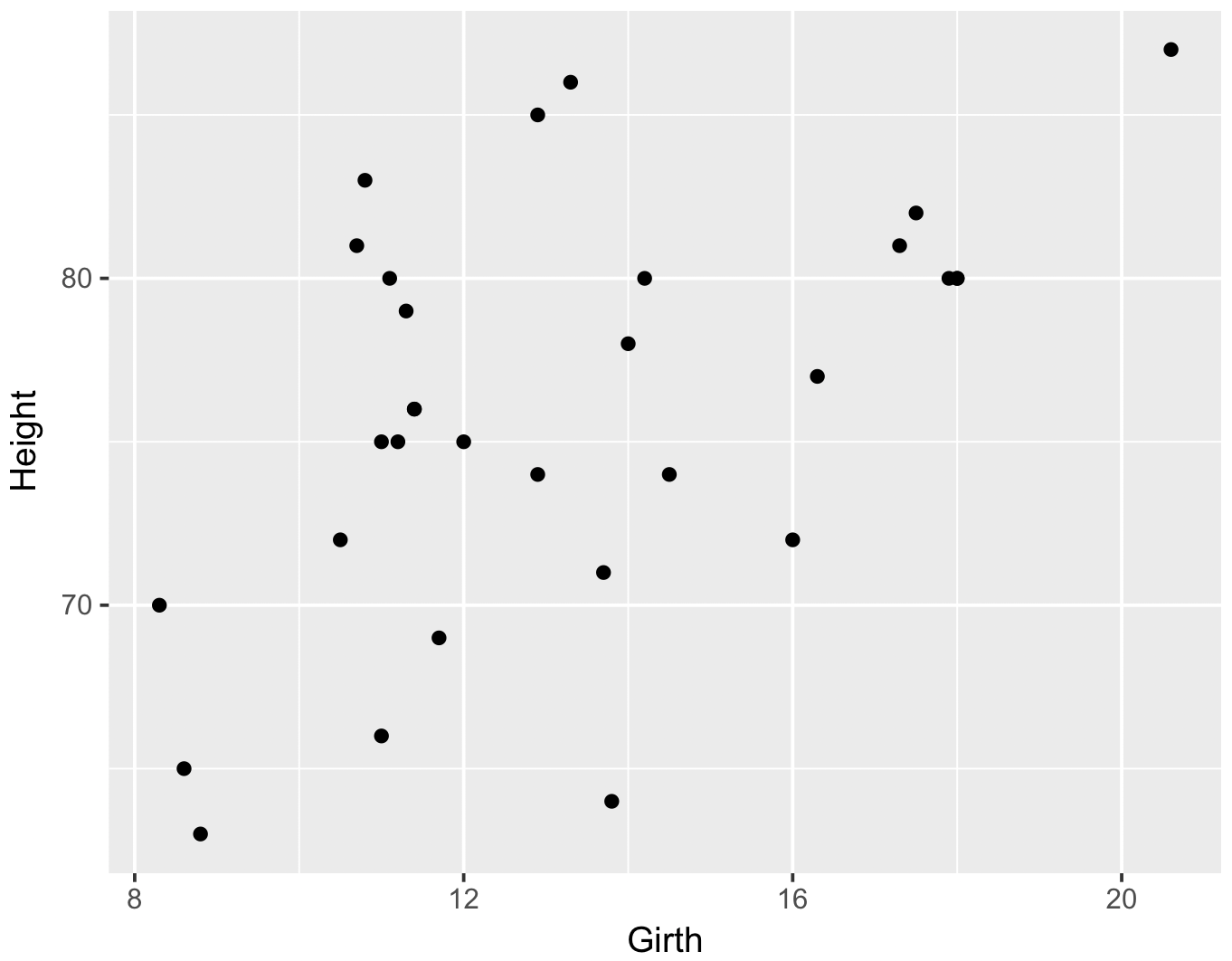
Stan コードによるモデル記述
単回帰モデルは、応答変数 y を説明変数 x で説明するモデルである。y と x の関係は次の関係がたり立つ。
\[ y = \mathcal{N}\left(\mu, \sigma \right)\] \[ \mu = \beta_{1}x + \beta_{0} \]このモデルの入力データは x (Girth) と y (Height) であり、パラメーターは β0、β1 および σ である。x および y は、後から与える入力データであるので data ブロックで定義する。β0、β1 および σ は、これから MCMC サンプリングを通して推定したいパラメーターであるので、parameters ブロックで定義する。また、回帰モデルにおける信頼区間および予測区間を計算するために、推定されたパラメーター(β0、β1 および σ)を用いて、Girth が 5-30 の範囲にあるときの木の高さをサンプリングするコードを generated quantities ブロックに記述する。
// lm.stan
data {
int N;
vector[N] x;
vector[N] y;
int new_N;
vector[new_N] new_x;
}
parameters {
real beta_0;
real beta_1;
real<lower=0> sigma;
}
transformed parameters {
real mu[N];
for (i in 1:N) {
mu[i] = beta_1 * x[i] + beta_0;
}
}
model {
for (i in 1:N) {
y[i] ~ normal(mu[i], sigma);
}
}
generated quantities {
real yhat[new_N];
real muhat[new_N];
for (i in 1:new_N) {
muhat[i] = beta_1 * new_x[i] + beta_0;
yhat[i] = normal_rng(muhat[i], sigma);
}
}パラメーター推定
R からデータを Stan モデルに代入するとき、R でデータをリストとして用意する必要がある。Stan コードで記述された data ブロックには、x、y、N、new_x、new_N の変数が定義されている。R で準備するリストにもこれらの名前をつける必要がある。データを準備できたら、これを stan 関数に代入してパラメーター推定を行う。stan 関数が実行されると、MCMC サンプリング結果が返される。この結果に、parameters および generated quantities ブロックで定義されているパラメーターはの事後分布が含まれる。
library(rstan)
d <- list(x = trees$Girth, y = trees$Height, N = nrow(trees),
new_x = seq(5, 30, 0.1), new_N = length(seq(5, 30, 0.1)))
fit <- stan(file = 'lm.stan', data = d)
fit
## Inference for Stan model: lm.
## 4 chains, each with iter=2000; warmup=1000; thin=1;
## post-warmup draws per chain=1000, total post-warmup draws=4000.
##
## mean se_mean sd 2.5% 25% 50% 75% 97.5% n_eff Rhat
## beta_0 62.07 0.12 4.54 52.83 59.10 62.14 65.01 71.07 1516 1
## beta_1 1.05 0.01 0.33 0.39 0.84 1.04 1.27 1.71 1511 1
## sigma 5.79 0.02 0.81 4.46 5.20 5.70 6.28 7.56 1643 1
## yhat[1] 67.19 0.12 6.59 53.71 62.94 67.26 71.54 80.32 3177 1
## yhat[2] 67.54 0.12 6.56 54.67 63.20 67.52 71.82 80.49 3224 1
## yhat[3] 67.65 0.12 6.54 54.79 63.25 67.64 71.90 80.39 3186 1
## ...
## yhat[248] 93.30 0.17 7.96 77.51 88.01 93.41 98.63 108.75 2304 1
## yhat[249] 93.32 0.16 8.10 76.58 87.98 93.61 98.73 108.88 2465 1
## yhat[250] 93.70 0.16 7.98 78.19 88.33 93.88 99.15 108.94 2537 1
## yhat[251] 93.74 0.16 8.16 77.65 88.26 93.76 99.21 109.42 2561 1
## ...
## muhat[1] 67.11 0.09 3.16 60.95 65.08 67.07 69.14 73.27 1125 1
## muhat[2] 67.22 0.09 3.13 61.12 65.21 67.18 69.22 73.33 1127 1
## muhat[3] 67.33 0.09 3.09 61.30 65.34 67.29 69.31 73.37 1129 1
## muhat[4] 67.44 0.09 3.06 61.46 65.47 67.41 69.40 73.40 1131 1
## ...
## muhat[249] 93.81 0.18 5.98 82.25 89.97 93.88 97.70 105.39 1078 1
## muhat[250] 93.92 0.18 6.02 82.28 90.05 93.98 97.84 105.56 1078 1
## muhat[251] 94.03 0.18 6.05 82.31 90.12 94.08 97.96 105.74 1078 1
## lp__ -67.48 0.05 1.38 -70.85 -68.11 -67.10 -66.50 -65.96 851 1fit 変数にはパラメーター推定時の設定条件およびパラメーターの推定結果が記録されている。例えば、この場合、4 つの chain で、それぞれ 2000 回のサンプリングが行われたことがわかる。また、2000 回のサンプリングのうち、最初の 1000 回はウォーミングアップのステップで、パラメーター推定時に使われていない。また、thin は間引きの間隔を表し、thin=1 の場合は、間引きが行われていなく、サンプリング結果をすべてを用いてパラメーター推定が行われる。例えば、thin=2 の場合、サンプリング結果のうちパラメーター推定に使用するのは 1 個おきとなる。
推定されたパラメーターの要約統計量は結果の中間部分に表示される。上の例では、beta_0、beta_1、sigma が推定されたパラメーターの要約統計量である。また、muhat および yhat は、generated quantities ブロックで再サンプリングされた値の要約統計量である。これらのサンプリング結果は extract 関数を使用して、行列または配列の形で取得できる。
ベイズ信用区間
信用区間とは、あるパラメーター(例えば母平均)の事後分布において、真の値が 1-α % の確率で含まれる区間である。Stan でモデルを構築したときに generated quantities ブロックで母平均 muhat の事後分布をサンプリングしたので、このパラメーターを取得すれば、95% 信用区間を求めることができるようになる。
library(dplyr)
library(ggplot2)
ms <- rstan::extract(fit)
df <- data.frame(d$new_x, t(apply(ms$muhat, 2, quantile, probs = c(0.025, 0.500, 0.975))))
colnames(df) <- c('x', 'lower', 'median', 'upper')
g <- ggplot(df, aes(x = x))
g <- g + geom_ribbon(aes(ymin = lower, ymax = upper), fill = '#000000', alpha = 0.4)
g <- g + geom_line(aes(y = median))
g <- g + geom_point(data = trees, aes(x = Girth, y = Height))
g <- g + xlab('Girth') + ylab('Height')
print(g)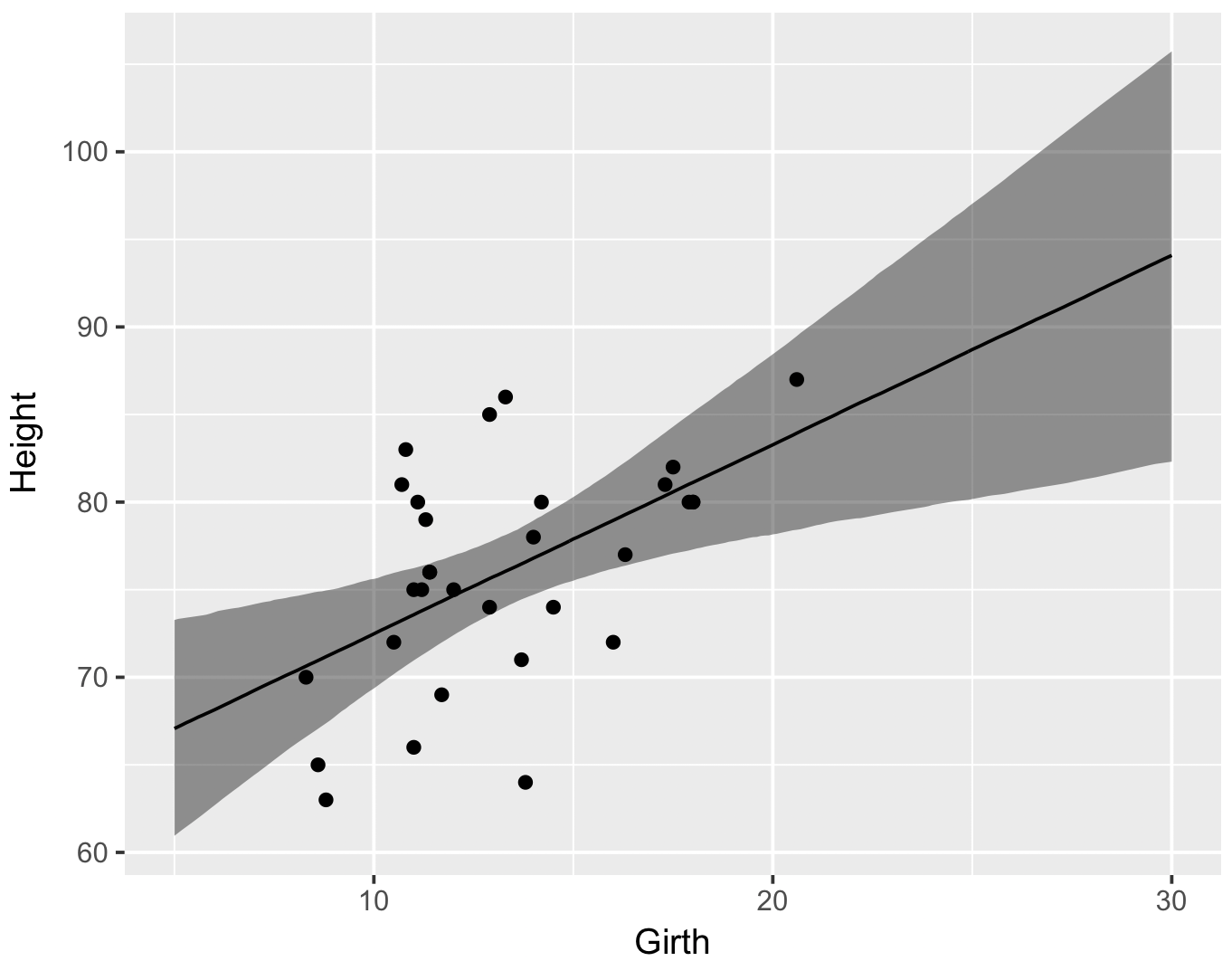
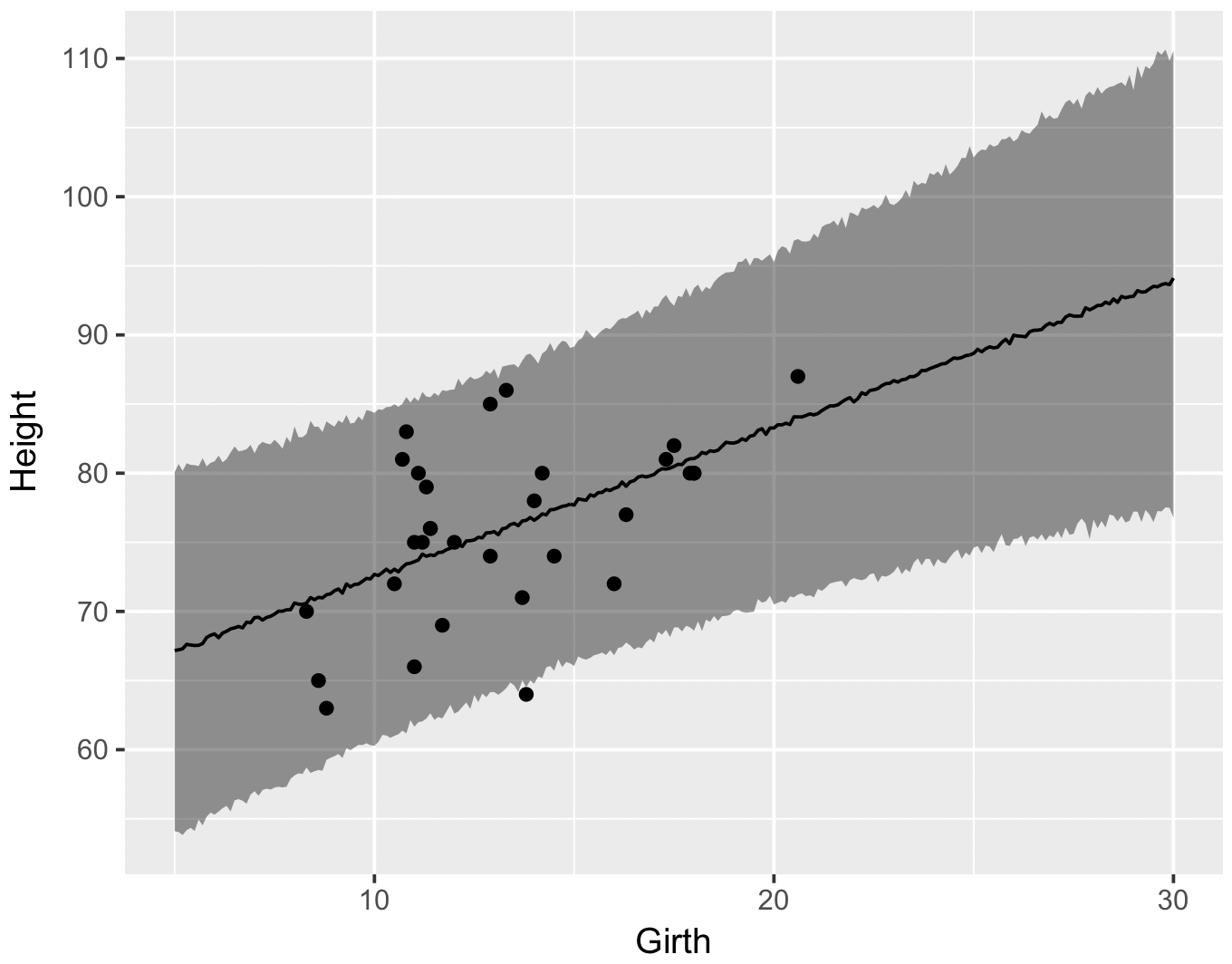
ベイズ予測区間
generated quantities ブロックでは、5-30 までの Girth を与えて、Height をサンプリングするように記述した。このサンプリング結果が yhat に保存される。ここで、extract 関数を用いて yhat のデータを取り出して、95% 予測区間を求める。
df <- data.frame(d$new_x, t(apply(ms$yhat, 2, quantile, probs = c(0.025, 0.500, 0.975))))
colnames(df) <- c('x', 'lower', 'median', 'upper')
g <- ggplot(df, aes(x = x))
g <- g + geom_ribbon(aes(ymin = lower, ymax = upper), fill = '#000000', alpha = 0.4)
g <- g + geom_line(aes(y = median))
g <- g + geom_point(data = trees, aes(x = Girth, y = Height))
g <- g + xlab('Girth') + ylab('Height')
print(g)