単回帰や重回帰は、一般化線形モデルにおいて誤差構造が正規分布で、リンク関数が恒等関数のときのことを表す。単回帰や重回帰は、基礎統計などでは lm 関数を使ってモデル構築が行われるが、一般化線形モデルを構築する際に使用する glm 関数を使用することもできる。このページでは、glm 関数を利用した回帰分析を紹介する。
単回帰分析
単回帰分析は、独立変数 X と従属変数 Y があるとき、Y を X で説明しようとする解析手法である。具体的に、2 つの変数を、Y = aX + b でモデルを立て、Y と X の関係を係数 a と b 用いて説明しようとする。ただし、従属変数 Y は正規分布に従う必要がある。
次は、sleep in mammals のデータに対して、glm 関数で単回帰分析を行う例である。sleep in mammals データは、種名(Species)、体重(BodyWt)、睡眠時間(TotalSleep)や寿命(LifeSpan)などが含むが、ここでは、寿命が睡眠時間のデータだけを使って、睡眠時間が寿命に与える影響を調べることにする。
x <- read.table("https://stats.biopapyrus.jp/data/sleep_in_mammals.txt", header = TRUE)
LifeSpan <- x$LifeSpan
TotalSleep <- x$TotalSleep
m <- glm(LifeSpan ~ TotalSleep, family = gaussian)
summary(m)
##
## Call:
## glm(formula = LifeSpan ~ TotalSleep, family = gaussian)
##
## Deviance Residuals:
## Min 1Q Median 3Q Max
## -22.075 -10.699 -2.000 6.841 76.190
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 36.9326 5.7696 6.401 4.45e-08 ***
## TotalSleep -1.6403 0.5057 -3.243 0.00207 **
## ---
## Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
##
## (Dispersion parameter for gaussian family taken to be 299.9632)
##
## Null deviance: 18754 on 53 degrees of freedom
## Residual deviance: 15598 on 52 degrees of freedom
## (8 observations deleted due to missingness)
## AIC: 465.21
##
## Number of Fisher Scoring iterations: 2summary によって出力される各項の意味は次のようになっている。
| Deviance Residuals | 残差平方和の四分位数 |
| Coefficients | 予測したパラメーターの値と統計量など |
| Estimate | モデルパラメーターの予測値 |
| Std. Error | 標準偏差 |
| t value | そのパラメーターが 0 かどうかの検定結果である統計量 |
| P(>|t|) | t value に対応する p value |
| Null deviance | Null Deviance = 2(LogLikelihood(飽和モデル) - LogLikelihood(帰無モデル)) によって計算される。飽和モデルは、データの個数分だけのパラメーター(回帰係数)を用意し、構築した回帰モデルである。帰無モデルは、パラメーター(回帰係数)を 1 つだけで構築した回帰モデルである。 |
| Residual deviance | Residual Deviance = 2(LogLikelihood(飽和モデル) - LogLikelihood(提案モデル)) によって計算される。提案モデルは、glm 関数に入力した回帰モデルである。上の例では、aX + b であるから、パラメーター(回帰係数)として 2 つある。 |
| AIC | 赤池情報量規準 |
plot(TotalSleep, LifeSpan)
abline(a = m$coefficients[1], b = m$coefficients[2])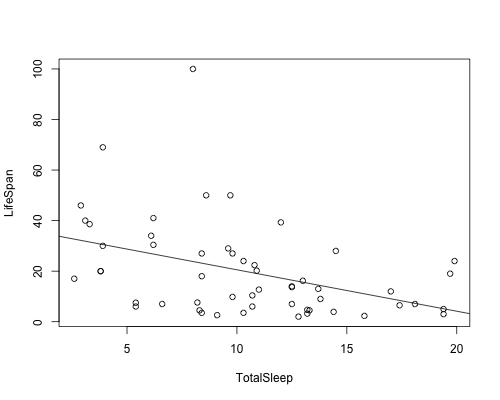
重回帰分析
glm 関数を利用した重回帰分析も、基本的に lm 関数と同じように行える。sleep in mammals のデータに対して、glm 関数で重回帰分析を行ってみる。sleep in mammals データには、種名(Species)、体重(BodyWt)、睡眠時間(TotalSleep)や寿命(LifeSpan)などが含まれている。ここで、寿命が睡眠時間と体重によって、影響されるかどうかを調べてみる。
x <- read.table("https://stats.biopapyrus.jp/data/sleep_in_mammals.html", header = TRUE)
LifeSpan <- x$LifeSpan
TotalSleep <- x$TotalSleep
BodyWt <- x$BodyWt
TotalSleep.std <- scale(TotalSleep)
BodyWt.std <- scale(BodyWt)
m <- glm(LifeSpan ~ TotalSleep.std + BodyWt.std, family = gaussian)
summary(m)
##
## Call:
## glm(formula = LifeSpan ~ TotalSleep.std + BodyWt.std, family = gaussian)
##
## Deviance Residuals:
## Min 1Q Median 3Q Max
## -20.114 -10.484 -2.591 6.490 77.322
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 19.630 2.333 8.415 3.28e-11 ***
## TotalSleep.std -6.476 2.422 -2.674 0.0101 *
## BodyWt.std 3.366 2.313 1.455 0.1518
## ---
## Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
##
## (Dispersion parameter for gaussian family taken to be 293.6548)
##
## Null deviance: 18754 on 53 degrees of freedom
## Residual deviance: 14976 on 51 degrees of freedom
## (8 observations deleted due to missingness)
## AIC: 465.01
##
## Number of Fisher Scoring iterations: 2