簡単なボックスプロット
ボックスプロットは、matplotlib ライブラリーの boxplot メソッドを利用して作成する。もっとも簡単な方法として、ボックスプロットにしたいデータをリストに保存し、そのリストを boxplot メソッドに渡す。複数項目ある場合は、リストのリストとして与える。x 軸のラベルは labels オプションで指定する。
import numpy as np
from matplotlib import pyplot as plt
import seaborn as sns
sns.set()
sns.set_style('whitegrid')
sns.set_palette('gray')
np.random.seed(2018)
x1 = np.random.normal(10, 2, 20)
x2 = np.random.normal(15, 3, 20)
x3 = np.random.normal(5, 1, 20)
fig = plt.figure()
ax = fig.add_subplot(1, 1, 1)
ax.boxplot([x1, x2, x3], labels=['leaf', 'stem', 'root'])
ax.set_xlabel('tissue')
ax.set_ylabel('dry weight [g]')
ax.set_ylim(0, 20)
plt.show()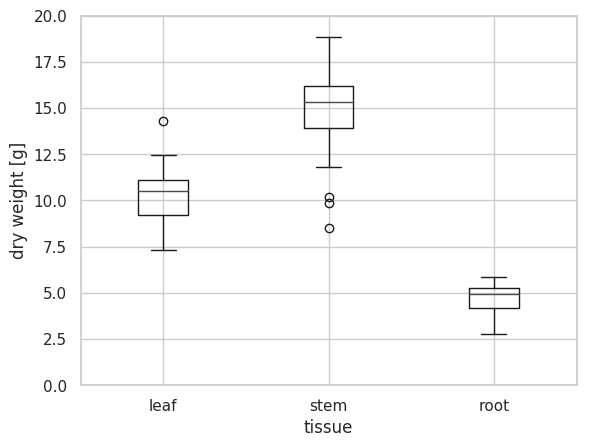
ボックスプロットから、データの四分位点などをすぐに確認できる。しかし、データの実際の値を確認できないといった欠点がある。そのため、現在では、ボックスプロットの上に、jitter プロットを重ねるよなグラフも見られるようになった。jitter プロットは、matplotlib 標準のメソッドを使って描くのは難しいので、ここでは seaborn ライブラリー中の stripplot メソッドを使う。
import numpy as np
import pandas as pd
from matplotlib import pyplot as plt
import seaborn as sns
sns.set()
sns.set_style('whitegrid')
sns.set_palette('Set3')
np.random.seed(2018)
df = pd.DataFrame({
'leaf': np.random.normal(10, 2, 20),
'stem': np.random.normal(15, 3, 20),
'root': np.random.normal(5, 1, 20)
})
df_melt = pd.melt(df)
print(df_melt.head())
## variable value
## 0 leaf 9.446465
## 1 leaf 11.163702
## 2 leaf 14.296799
## 3 leaf 7.441026
## 4 leaf 11.004554
fig = plt.figure()
ax = fig.add_subplot(1, 1, 1)
sns.boxplot(x='variable', y='value', data=df_melt, showfliers=False, ax=ax)
sns.stripplot(x='variable', y='value', data=df_melt, jitter=True, color='black', ax=ax)
plt.show()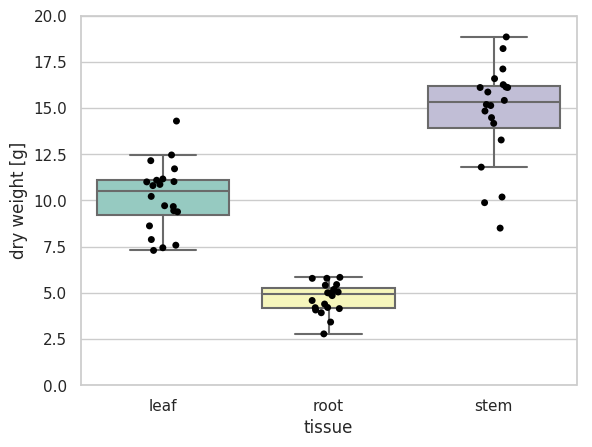
横に並べたボックスプロット
複数項目の場合は、次のように、データを一つのデータフレームにまとめてから、seaborn ライブラリーの boxplot メソッドを使うと簡単に描ける。
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
plt.style.use('default')
sns.set()
sns.set_style('whitegrid')
sns.set_palette('Set3')
np.random.seed(2018)
# data of Ta (T. aestivum) group
dfTa = pd.DataFrame({
'leaf': np.random.normal(10, 2, 20),
'stem': np.random.normal(15, 3, 20),
'root': np.random.normal(5, 1, 20)
})
# data of At (A. tauschii) group
dfAt = pd.DataFrame({
'leaf': np.random.normal(12, 3, 20),
'stem': np.random.normal(12, 3, 20),
'root': np.random.normal(6, 2, 20)
})
# merge the two data frames to one data frame
dfTa_melt = pd.melt(dfTa)
dfTa_melt['species'] = 'T. aestivum'
dfAt_melt = pd.melt(dfAt)
dfAt_melt['species'] = 'A. tauschii'
df = pd.concat([dfAt_melt, dfTa_melt], axis=0)
print(df.head())
## variable value species
## 0 leaf 11.871913 A. tauschii
## 1 leaf 16.013749 A. tauschii
## 2 leaf 11.930841 A. tauschii
## 3 leaf 14.794698 A. tauschii
## 4 leaf 8.642995 A. tauschii
fig = plt.figure()
ax = fig.add_subplot(1, 1, 1)
sns.boxplot(x='variable', y='value', data=df, hue='species', palette='Dark2', ax=ax)
ax.set_xlabel('tissue')
ax.set_ylabel('dry weight [g]')
ax.set_ylim(0, 20)
ax.legend()
plt.show()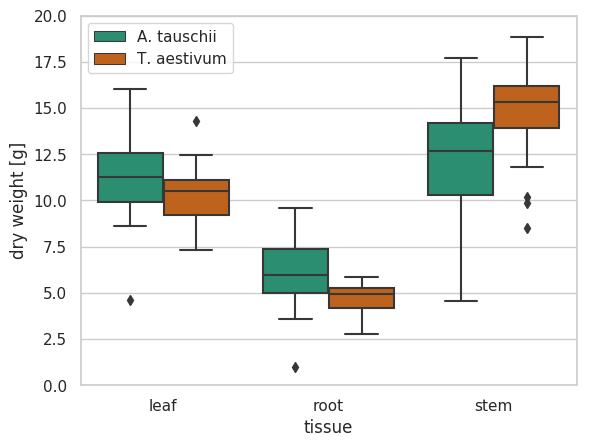
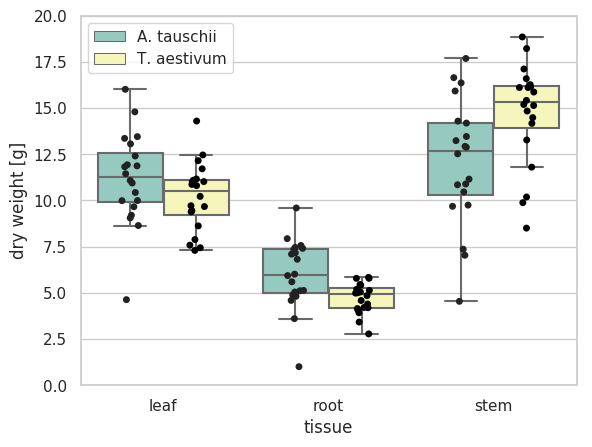
ボックスプロットが複数個横に並んでも、jitter プロットを追加できる。
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
plt.style.use('default')
sns.set()
sns.set_style('whitegrid')
sns.set_palette('Set3')
np.random.seed(2018)
# data of Ta (T. aestivum) group
dfTa = pd.DataFrame({
'leaf': np.random.normal(10, 2, 20),
'stem': np.random.normal(15, 3, 20),
'root': np.random.normal(5, 1, 20)
})
# data of At (A. tauschii) group
dfAt = pd.DataFrame({
'leaf': np.random.normal(12, 3, 20),
'stem': np.random.normal(12, 3, 20),
'root': np.random.normal(6, 2, 20)
})
dfTa_melt = pd.melt(dfTa)
dfTa_melt['species'] = 'T. aestivum'
dfAt_melt = pd.melt(dfAt)
dfAt_melt['species'] = 'A. tauschii'
df = pd.concat([dfAt_melt, dfTa_melt], axis=0)
print(df.head())
## variable value species
## 0 leaf 11.871913 A. tauschii
## 1 leaf 16.013749 A. tauschii
## 2 leaf 11.930841 A. tauschii
## 3 leaf 14.794698 A. tauschii
## 4 leaf 8.642995 A. tauschii
fig = plt.figure()
ax = fig.add_subplot(1, 1, 1)
sns.boxplot(x='variable', y='value', data=df, hue='species', showfliers=False, palette='Set3', ax=ax)
sns.stripplot(x='variable', y='value', data=df, hue='species', dodge=True, jitter=True, color='black', ax=ax)
handles, labels = ax.get_legend_handles_labels()
# 4 legends for defaul: boxplot x 2 species, and stripplot x 2 species
# use the first 2 legends
ax.legend(handles[0:2], labels[0:2])
ax.set_xlabel('tissue')
ax.set_ylabel('dry weight [g]')
ax.set_ylim(0, 20)
plt.show()