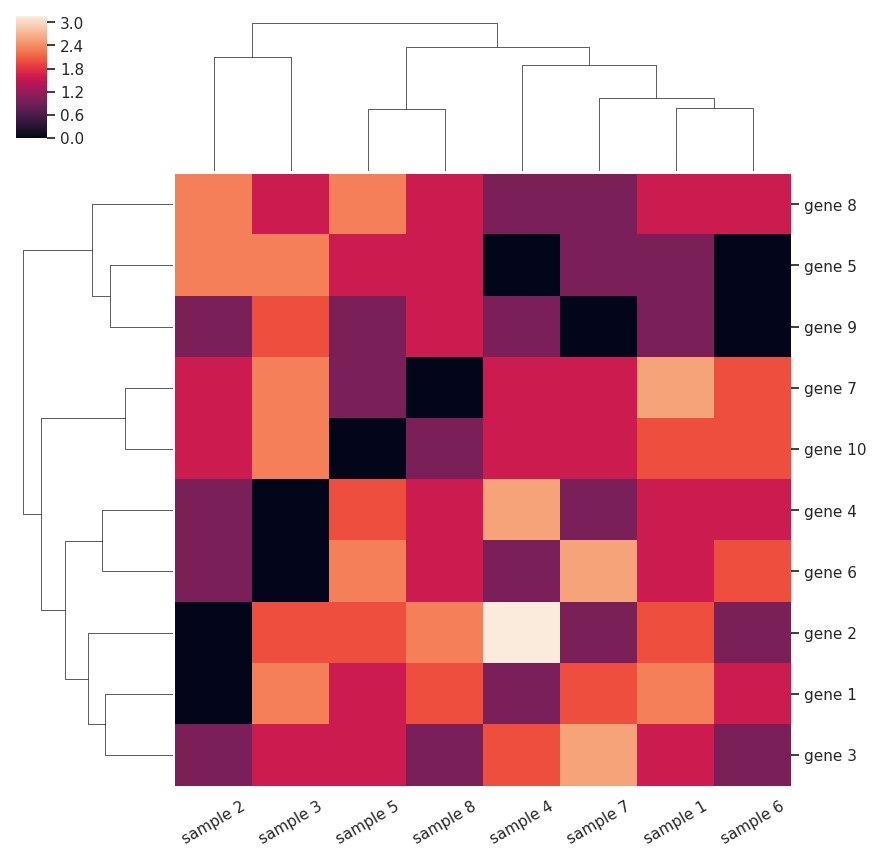
ヒートマップを描く seaborn のメソッドとして heatmap と clustermap がある。heatmap はヒートマップを描くだけであるのに対して、clustermap は縦と横の項目をクラスタリングさせてからヒートマップを描く。クラスタリングの結果は、ヒートマップの上あるいは左に樹形図として書き出される。クラスタリングのアルゴリズムは method オプションで指定し、要素同士の距離は metric オプションで指定する。また、clustermap で描き出されたヒートマップのラベルを回転させて見やすくするには plt.setp メソッドを利用する。
import numpy as np
import pandas as pd
import seaborn as sns
import matplotlib.pyplot as plt
np.random.seed(2018)
plt.style.use('default')
sns.set()
sns.set_style('whitegrid')
# make 10x8 matrix
data = np.random.binomial(100, 0.02, 80).reshape((10, 8))
data = np.log2(data + 1)
df = pd.DataFrame(data,
index=['gene ' + str(i + 1) for i in range(10)],
columns=['sample ' + str(i + 1) for i in range(8)])
print(df)
# haetmap
sns_plot = sns.clustermap(df, method='ward', metric='euclidean')
plt.setp(sns_plot.ax_heatmap.get_yticklabels(), rotation=0)
plt.setp(sns_plot.ax_heatmap.get_xticklabels(), rotation=30)
plt.show()
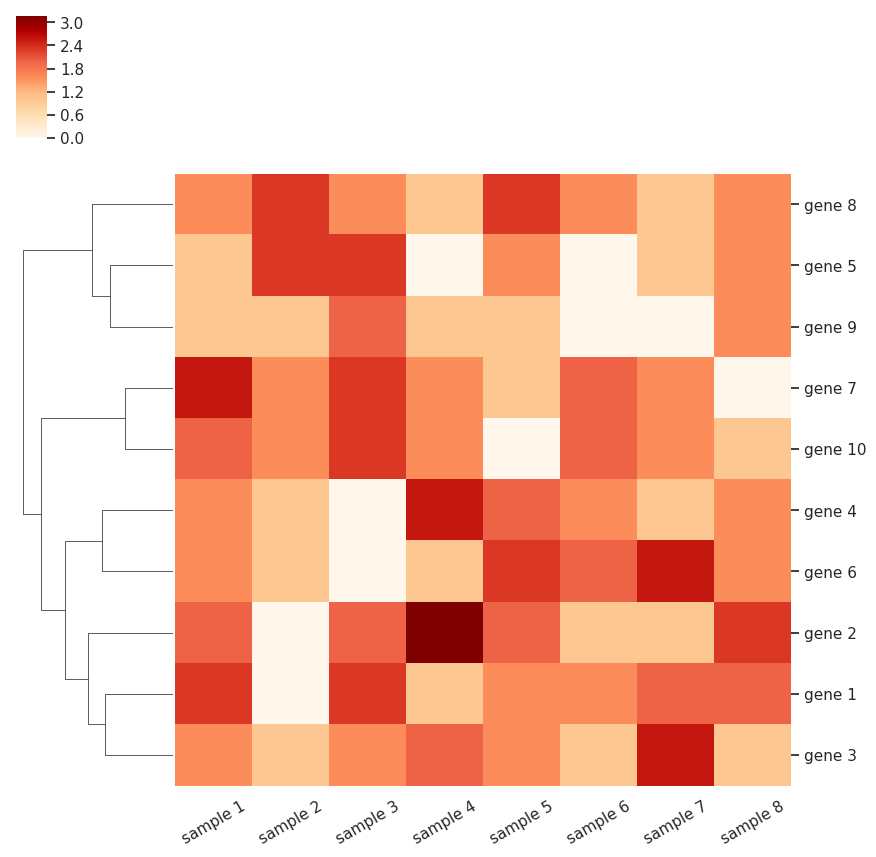
col_cluster = False または row_cluster = False を指定すると、列または行に対してクラスタリングが行われなくなる。また、heatmap メソッドと同様に、cmap オプションでカラーパレットを変更することができる。
sns_plot = sns.clustermap(df, method='ward', metric='euclidean',
col_cluster = False, cmap='OrRd')
plt.setp(sns_plot.ax_heatmap.get_yticklabels(), rotation=0)
plt.setp(sns_plot.ax_heatmap.get_xticklabels(), rotation=30)
plt.show()
linewidths オプションでタイルとタイルの間の余白を指定することができるようになる。
sns_plot = sns.clustermap(df, method='ward', metric='euclidean',
col_cluster = False, cmap='PuBu', linewidths=0.5)
plt.setp(sns_plot.ax_heatmap.get_yticklabels(), rotation=0)
plt.setp(sns_plot.ax_heatmap.get_xticklabels(), rotation=30)
plt.show()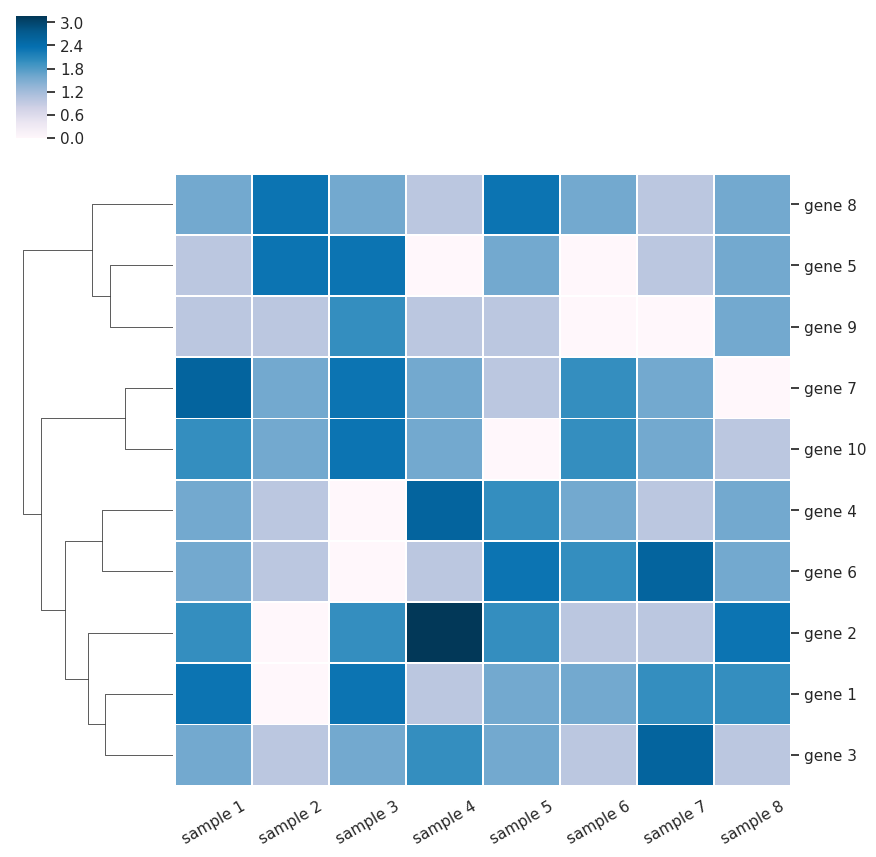
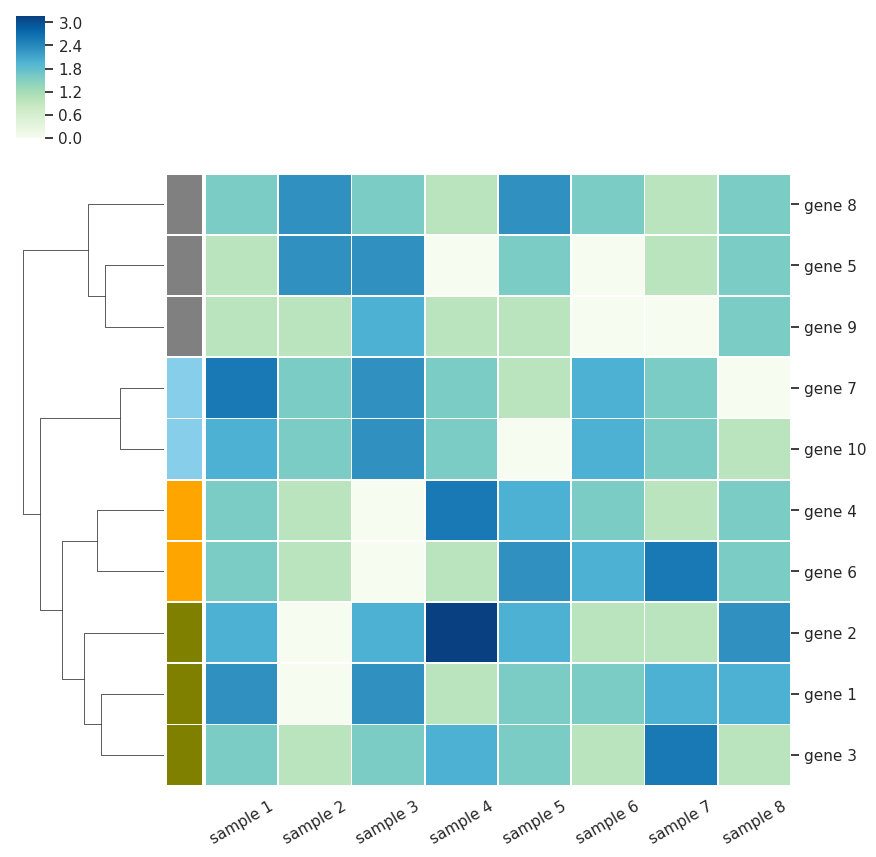
row_colors または col_colors オプションを使用することで、行方向あるいは列方向の樹形図の付け根の部分に色を塗ることができる。この際、クラスタリングの前の順序で、あらかじめ色のベクトルを作成しておく必要がある。
# set color for gene 1 to 10
gene_loc = ['olive', 'olive', 'olive', 'orange', 'gray',
'orange', 'skyblue', 'gray', 'gray', 'skyblue']
# haetmap
sns_plot = sns.clustermap(df, method='ward', metric='euclidean',
col_cluster = False, cmap='GnBu', linewidths=0.5,
row_colors = gene_loc)
plt.setp(sns_plot.ax_heatmap.get_yticklabels(), rotation=0)
plt.setp(sns_plot.ax_heatmap.get_xticklabels(), rotation=30)
plt.show()