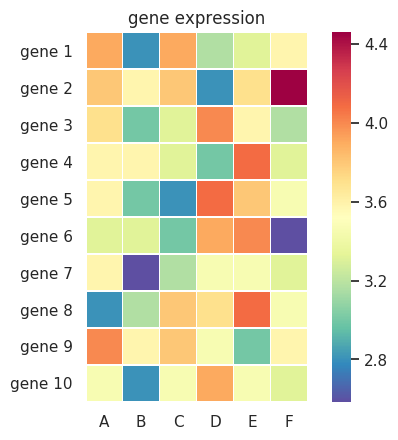
Python の matplotlib ライブラリーでもヒートマップを作成することは可能だが、seaborn ライブラリーの heatmap メソッドを用いたほうが簡単。ヒートマップで表したいデータは二次元配列の形で heatmap に与える。また、heatmap のオプションを square=True に指定すると、ヒートマップの各タイルが正方形で描かれるようになり、グラフ全体がきれいになる。
import numpy as np
import pandas as pd
import seaborn as sns
import matplotlib.pyplot as plt
np.random.seed(2018)
plt.style.use('default')
sns.set()
sns.set_style('whitegrid')
# make 10x6 matrix
data = np.random.binomial(100, 0.05, 60).reshape((10, 6))
data = np.log2(data + 1)
df = pd.DataFrame(data,
index=['gene ' + str(i + 1) for i in range(10)],
columns=['A', 'B', 'C', 'D', 'E', 'F'])
# haetmap
sns.heatmap(df, square=True)
plt.title('gene expression')
plt.show()
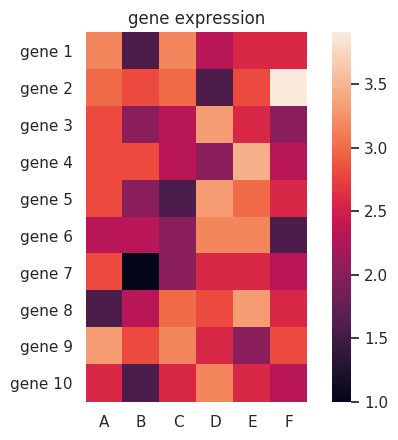
カラーパレットは cmap オプションで指定する。
# haetmap
sns.heatmap(df, square=True, cmap='RdYlGn_r')
plt.title('gene expression')
plt.show()
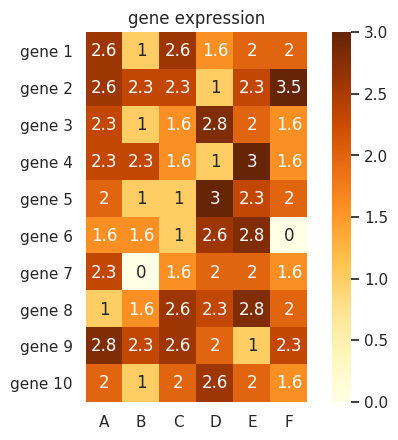
色のグラデーションについて最大値と最小値を指定することができる。例えば、次のようにすると、最大値 3 以上の値は 3 と同じ色で描かれる。また、annot=True とすることで、各タイルに実際の値が書き出される。
import numpy as np
import pandas as pd
import seaborn as sns
import matplotlib.pyplot as plt
np.random.seed(2018)
plt.style.use('default')
sns.set()
sns.set_style('whitegrid')
# make 10x6 matrix
data = np.random.binomial(100, 0.03, 60).reshape((10, 6))
data = np.log2(data + 1)
df = pd.DataFrame(data,
index=['gene ' + str(i + 1) for i in range(10)],
columns=['A', 'B', 'C', 'D', 'E', 'F'])
# haetmap
sns.heatmap(df, annot=True, square=True, cmap='YlOrBr', vmin=0, vmax=3)
plt.title('gene expression')
plt.show()
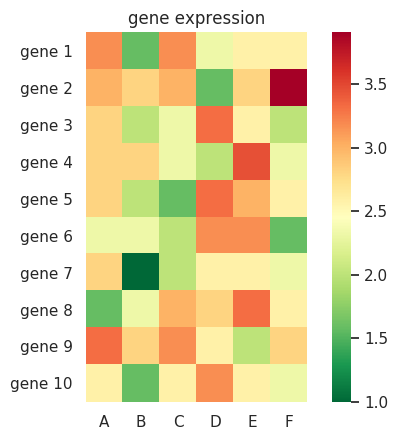
linewidths オプションでタイルとタイルの間の余白を指定することができるようになる。
import numpy as np
import pandas as pd
import seaborn as sns
import matplotlib.pyplot as plt
np.random.seed(2018)
plt.style.use('default')
sns.set()
sns.set_style('whitegrid')
# make 10x6 matrix
data = np.random.binomial(100, 0.1, 60).reshape((10, 6))
data = np.log2(data + 1)
df = pd.DataFrame(data,
index=['gene ' + str(i + 1) for i in range(10)],
columns=['A', 'B', 'C', 'D', 'E', 'F'])
# haetmap
sns.heatmap(df, square=True, cmap='Spectral_r', linewidths=0.5)
plt.title('gene expression')
plt.show()