circos プロットはゲノム構造の視覚化の際によく利用されるプロットである。circlize (Gu et al., 2014)、RCircos (Zhang et al., 2013) や OmicCircos (Hu et al., 2014) などのパッケージを利用して描くことができる。
circlize パッケージ
circlize パッケージを用いて circos プロットを描く場合、以下の手順で行う。
circos.initialize関数を用いて、円の分割などの circos プロットのレイアウトの初期設定を行う。circos.points、circos.linesなどの基本関数を利用して、circos プロット上の特定領域に点グラフや線グラフを描く。circ.clear関数を実行して、描画を終わらせる。
まず、パッケージをロードしてから、乱数によりデータフレーム型のサンプルデータを作成する。
library(circlize)
n <- 1000
d <- data.frame(
chr = sample(c("chr1", "chr2", "chr3", "chr4", "chr5", "chr6"), n, replace = TRUE),
x = rnorm(n),
y = rnorm(n)
)
head(d)
## chr x y
## 1 chr4 0.1361047 -0.9054468
## 2 chr6 1.3881900 0.2430469
## 3 chr3 0.6885233 -1.8838541
## 4 chr2 -0.5461639 -0.1173320
## 5 chr5 -0.7981908 0.4724849
## 6 chr5 -0.2729639 1.0990361circos プロットの初期化を行い、点プロットを追加する。
circos.initialize(factors = d$chr, x = d$x)
circos.trackPlotRegion(factors = d$chr, y = d$y, panel.fun = function(x, y) {
circos.axis()
})
circos.trackPoints(d$chr, d$x, d$y, pch = 16, cex = 0.5, col = 1:6)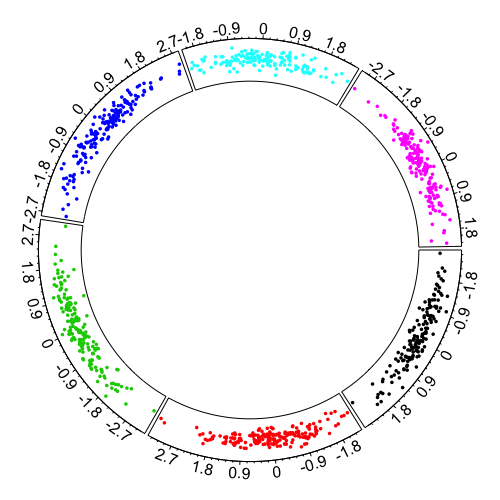
次に、円の内側に新しいトラックを追加し、先ほどの d$x の分布をヒストグラムで描く。
circos.trackHist(d$chr, d$x)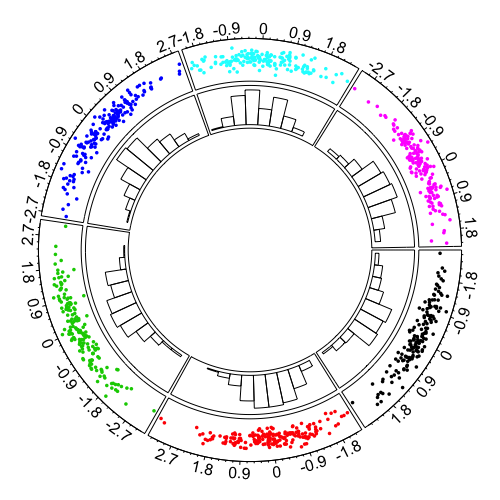
リンクをつなげる。
circos.link("chr1", 0, "chr2", 0)
circos.link("chr4", c(-2, 0), "chr3", c(-2, 2), col = "blue")
circos.link("chr4", c(0, 2), "chr6", c(-2, 0), col = "orange")
circos.link("chr5", c(0, 2), "chr1", c(-2, 2), col = "lightgrey")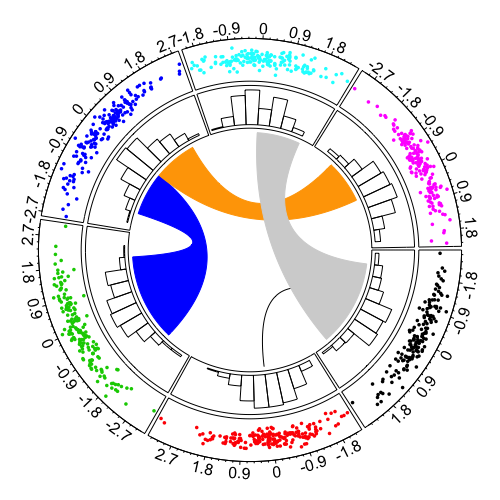
References
- circlize Implements and enhances circular visualization in R. Bioinformatics. 2014, 30(19):2811-2. DOI: 10.1093/bioinformatics/btu393 PMID: 24930139
- RCircos: an R package for Circos 2D track plots. BMC Bioinformatics. 2013, 14:244. DOI: 10.1186/1471-2105-14-244 PMID: 23937229
- OmicCircos: A Simple-to-Use R Package for the Circular Visualization of Multidimensional Omics Data. Cancer Inform. 2014, 13:13-20. DOI: 10.4137/CIN.S13495 PMID: 24526832