折れ線を描くとき lines 関数を利用すると便利である。
折れ線グラフ
折れ線グラフ
plot 関数を利用して、描画キャンパス(デバイス)を用意して、lines 関数で折れ線グラフを描く。まず、次のようなサンプルデータを用意する。
x <- data.frame(
ColdTreat = c(2.2, 2.1, 2.0, 2.0, 1.8, 1.9, 1.8, 1.5),
WarmTreat = c(2.1, 2.2, 2.3, 2.5, 2.6, 2.6, 2.8, 3.0),
Control = c(2.0, 2.1, 2.0, 2.2, 2.1, 2.2, 2.0, 2.1)
)
head(x)
## ColdTreat WarmTreat Control
## 1 2.2 2.1 2.0
## 2 2.1 2.2 2.1
## 3 2.0 2.3 2.0
## 4 2.0 2.5 2.2
## 5 1.8 2.6 2.1
## 6 1.9 2.6 2.2次に、グラフを描いていく。
xrange <- 1:nrow(x)
plot(0, 0, type = "n", xlim = range(xrange), ylim = range(0, x), xlab = "time", ylab = "weight")
cols <- c("black", "red", "blue")
ltys <- c("solid", "dotted", "dashed")
for (i in 1:ncol(x)) {
lines(xrange, x[, i], col = cols[i], lty = ltys[i])
}
legend("bottomleft", legend = colnames(x), col = cols, lty = ltys)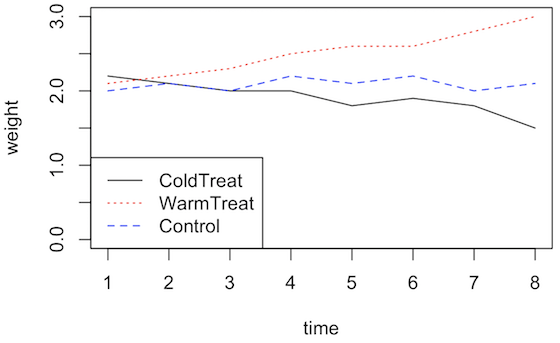
折れ線グラフとエラーバー
生物実験などで、複製実験などがあるときに、各点において標準偏差を計算することができる。その標準偏差をエラーバーとして、平均値の折れ線グラフに書き加えることができる。以下に ColdTreat、WarmTreat、Control の 3 群それぞれに 3 つの複製実験が行われたと仮定して、折れ線グラフとエラーバーを描く方法を示す。
cold.treat <- data.frame(
bio.rep.1 = c(2.2, 2.1, 2.0, 2.0, 1.8, 1.9, 1.8, 1.5),
bio.rep.2 = c(2.1, 2.1, 1.9, 1.8, 1.7, 1.7, 1.5, 1.4),
bio.rep.3 = c(2.2, 2.0, 1.9, 1.9, 1.8, 1.7, 1.6, 1.5)
)
cold.treat <- data.frame(
bio.rep.1 = c(2.2, 2.1, 2.0, 2.0, 1.8, 1.9, 1.8, 1.5),
bio.rep.2 = c(2.1, 2.1, 1.9, 1.8, 1.7, 1.7, 1.5, 1.4),
bio.rep.3 = c(2.2, 2.0, 1.9, 1.9, 1.8, 1.7, 1.6, 1.5)
)
warm.treat <- data.frame(
bio.rep.1 = c(2.1, 2.2, 2.3, 2.5, 2.6, 2.6, 2.8, 3.0),
bio.rep.2 = c(2.2, 2.3, 2.4, 2.5, 2.6, 2.7, 2.8, 3.1),
bio.rep.3 = c(2.2, 2.3, 2.3, 2.4, 2.5, 2.6, 2.7, 3.0)
)
control <- data.frame(
bio.rep.1 = c(2.0, 2.1, 2.0, 2.2, 2.1, 2.2, 2.0, 2.1),
bio.rep.2 = c(2.2, 2.2, 2.1, 2.2, 2.1, 2.0, 2.1, 2.2),
bio.rep.3 = c(2.1, 2.1, 2.0, 2.1, 2.2, 2.1, 2.0, 2.1)
)平均と標準偏差を計算する。
x.mean <- data.frame(
ColdTreat = apply(cold.treat, 1, mean),
WarmTreat = apply(warm.treat, 1, mean),
Control = apply(control, 1, mean)
)
x.sd <- data.frame(
ColdTreat = apply(cold.treat, 1, sd),
WarmTreat = apply(warm.treat, 1, sd),
Control = apply(control, 1, sd)
)平均値と標準偏差を計算した後に、平均値で折れ線グラフを描く。続いて、arrow 関数でエラーバーを追加する。
xrange <- 1:nrow(x.mean)
plot(0, 0, type = "n", xlim = range(xrange), ylim = range(0, c(x.mean + x.sd, x.mean - x.sd)), xlab = "time", ylab = "weight")
cols <- c("black", "red", "blue")
ltys <- c("solid", "dotted", "dashed")
for (i in 1:ncol(x.mean)) {
lines(xrange, x.mean[, i], col = cols[i], lty = ltys[i])
arrows(xrange, x.mean[, i] - x.sd[, i], xrange, x.mean[, i] + x.sd[, i], code = 3, lwd = 1, col = cols[i], angle = 90, length = 0.05)
}
legend("bottomleft", legend = colnames(x.mean), col = cols, lty = ltys)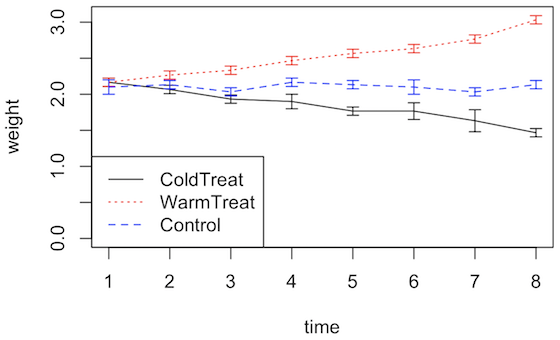
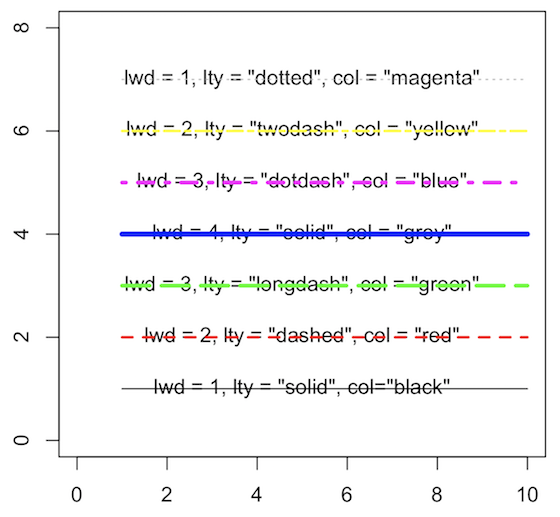
線のプロパティ
lines 関数を利用して折れ線グラフを描くとき、線の色、太さやタイプなどは col や lty などのオプションで指定できる。
plot(c(0,0), xlim=c(0,10), ylim = c(0,8), type = "n")
text(5, 1, 'lwd = 1, lty = "solid", col="black"')
lines(c(1, 10), c(1, 1), lwd = 1 , lty = "solid", col = "black")
text(5, 2, 'lwd = 2, lty = "dashed", col = "red"')
lines(c(1, 10), c(2, 2), lwd = 2, lty = "dashed", col = "red")
text(5, 3, 'lwd = 3, lty = "longdash", col = "green"')
lines(c(1, 10), c(3, 3), lwd = 3, lty = "longdash", col = "green")
text(5, 4, 'lwd = 4, lty = "solid", col = "grey"')
lines(c(1, 10), c(4, 4), lwd = 4, lty = "solid", col = "blue")
text(5, 5, 'lwd = 3, lty = "dotdash", col = "blue"')
lines(c(1, 10), c(5, 5), lwd = 3, lty = "dotdash", col = "magenta")
text(5, 6, 'lwd = 2, lty = "twodash", col = "yellow"')
lines(c(1, 10), c(6, 6), lwd = 2, lty = "twodash", col = "yellow")
text(5, 7, 'lwd = 1, lty = "dotted", col = "magenta"')
lines(c(1, 10), c(7, 7), lwd = 1, lty = "dotted", col = "grey")